耗時10年,人類染色體4D圖譜發佈:揭開DNA摺疊真相,探究不明遺傳病原因
上個月,人類染色體4D圖譜正式發佈,人類對生命之謎的理解又上了一個臺階。
如果說人類全基因組序列是一本超全通訊錄,摸清了每個基因叫什麼住哪裏,染色體4D圖譜計劃(4D Nucleome,以下簡稱4DN)就是一個信息量爆炸的朋友圈,記載了它們在細胞中是如何彼此聯繫,關鍵時刻又是如何協作的。
這項由人類研究自身的宏大工程啓動十年,終於迎來了階段性大成果。
它匯聚了普林斯頓大學、賓夕法尼亞大學、卡耐基梅隆大學等多所國際高校與研究機構的力量,也有我國復旦大學、浙江大學等團隊參與其中。最終,研究人員完成了這篇由90位作者共同署名的聯合論文,並於12月18日發表在頂級期刊《自然》(Nature)上。

論文作者名單| 圖源 Springer nature
在這項研究裏,研究人員系統地呈現了人體中的兩種代表性細胞——分化能力極強的人類胚胎幹細胞,以及到達了分化終點,完全成型了的成纖維細胞中的染色體三維結構。
同時,研究人員還引入了第四維度——時間,描述了隨着時間推移,細胞中的三維結構會如何變化。
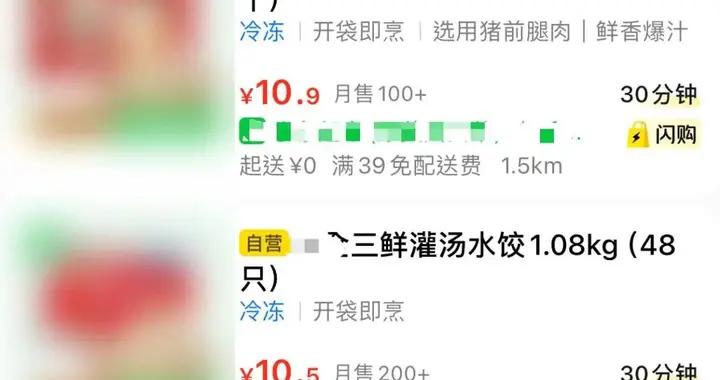
人類胚胎幹細胞(中央)和餵養它們的小鼠胚胎成纖維細胞(外周),這兩種細胞的形態、結構有着巨大的差異 | wikipedia commons
這項研究爲未來相關研究奠定了重要基礎,大大加深了我們對染色體三維結構的認識。
比如,它爲遺傳病診斷提供了新視角:一些機制不明的齶裂、脣裂,或多指、並指等新生兒發育異常,可能並非源於蛋白序列改變,而是染色體摺疊出錯。還有,更多目前病因不明的疾病也許能借助4DN的框架被重新解釋,我們對自身基因的認識又上升了一個緯度。
4DN計劃,讓基因序列多了兩個維度
在本世紀初,由全球多國科研機構合作完成的人類全基因組圖譜發佈了。這項計劃測出了人類基因組的完整序列,讓我們第一次清楚地知道每條染色體上鹼基的排列順序,以及基因大致分佈在哪裏。
但這份圖譜有一個巨大的缺陷,它只記錄了攤平的基因二維序列,但在真實的生理環境中,染色圖的結構要複雜得多。DNA在細胞核中會經歷多重摺疊,形成複雜的三維結構,這些結構還會隨着細胞狀態變化而改變,這是細胞核精確調控生理活動的基礎,僅憑鹼基序列是看不出來的。

染色體3D圖譜概念圖,很像乾脆面 |Springer nature
爲了進一步解釋DNA在空間和時間上的變化,4DN計劃誕生了。 它自2014年啓動,迄今已產出800餘篇論文,積累了大量數據、方法與工具。如今,這些成果正在匯聚成一份關鍵的公共資源:一張更接近全景的人類染色體4D圖譜。
我們在初中教課書上看到的像扭轉的梯子一樣的DNA示意圖,其實是2D的,只記錄了染色體上A、T、C、G四種鹼基序列的種類和順序。
常見的DNA示意圖|healthline
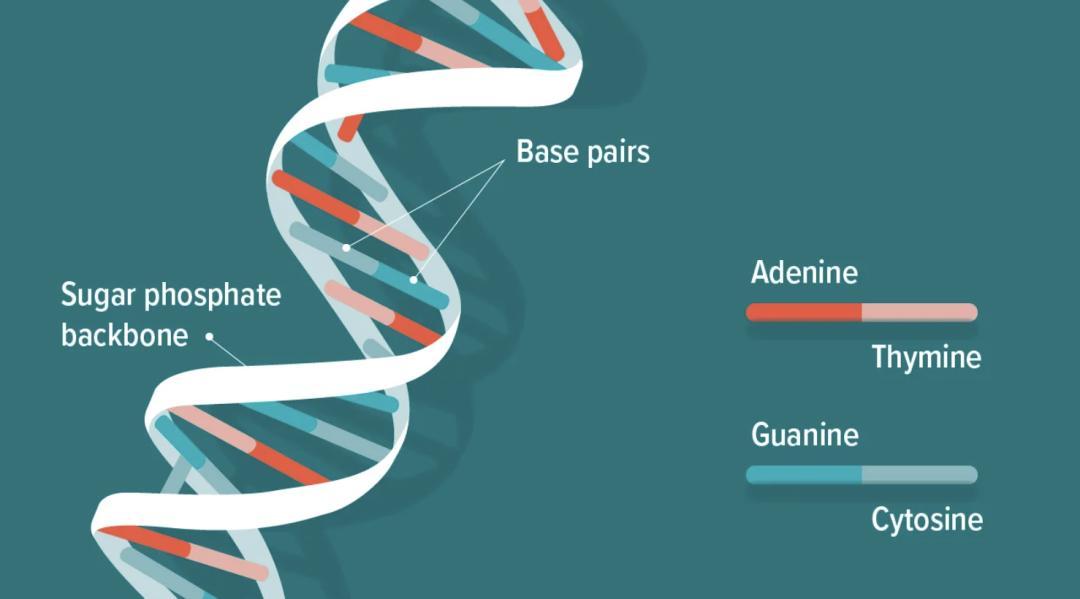
而在人的細胞內,基因組並非像手串一樣一條到底,事實上,基因組會進一步摺疊、堆疊、打包,形成毛線團一般層層嵌套的三維染色質與染色體結構。
層層摺疊的染色質,真實的情況遠比教科書上的複雜 |wikipedia commons
在這些三維結構中,研究人員尤其關注一種關鍵單元——染色質環(Chromatin loop)。染色質環與基因組功能緊密相關,理解這些環,等於在理解基因組裏遠距離調控的方式,讓三維結構能夠和細胞裏的真實生理功能對上號,二維平面的基因組就一下子立了起來。
基因表達並不是單點開關,一個基因能否啓動、何時啓動、強度多大,往往需要多個遠端調控元件協同決定。好比在工作中,啓動一個項目往往得湊齊好幾位領導的簽字。但爲了獲得領導的簽名,一般都要跑這跑那,而基因組中也是類似的。
目的基因和調控元件在二維,也就是基因序列上,可能隔着十萬八千里,幾十萬到上百萬個鹼基都有可能。要讓它們真正協同工作,就需要把 DNA 摺疊起來,讓原本相距遙遠的片段在空間裏靠得足夠近,於是染色質環就形成了。
爲了儘可能全面且精確地找出所有染色質環,研究人員使用了多種捕捉細胞核中染色質環的技術,對環進行多角度地識別和整合,繪製出了精確的染色體三維圖譜。
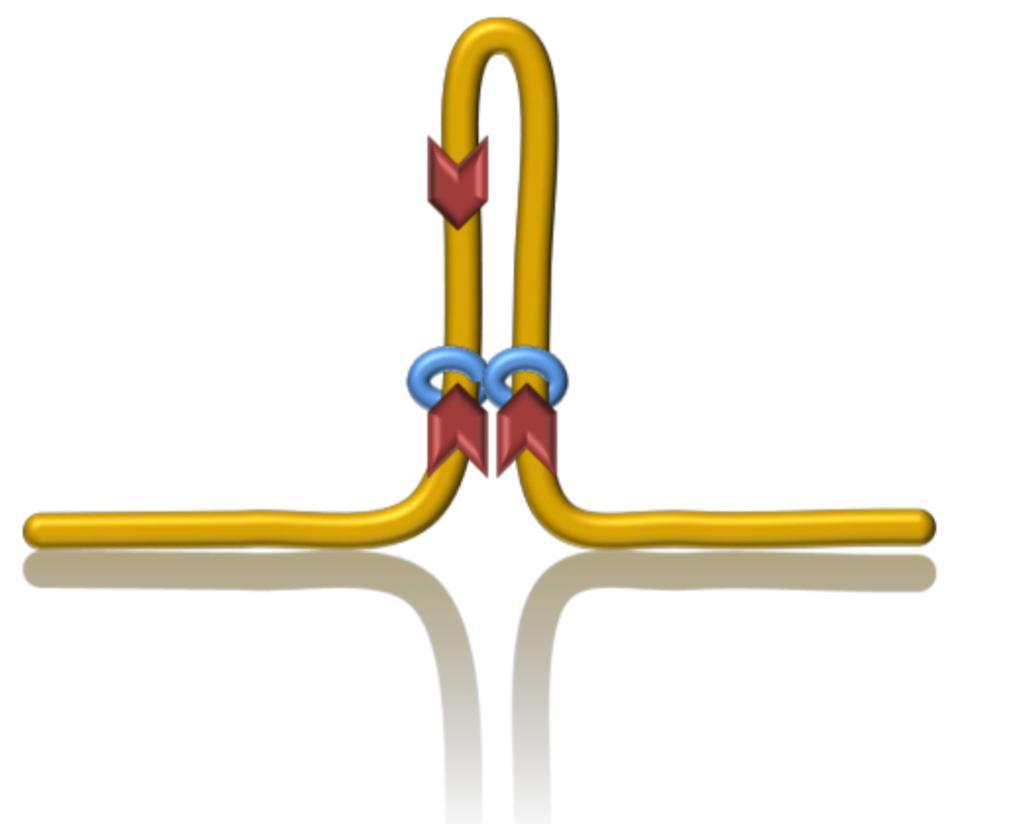
凝聚素使染色體成環 | wikipedia commons
到這裏,我們只是把基因組從二維升維到了三維。
但這個三維結構並非固定不變:隨着細胞週期推進、分化發生、外界刺激到來,染色體會改變摺疊方式與相互接觸的模式。科學家希望把這種結構隨時間或者狀態變化的維度也納入進來,這就是4D圖譜中的第四維——時間。
有了研究人員建立的圖譜,我們就能查到許多僅憑DNA序列本身看不出來的信息:例如哪些基因在三維空間裏會被摺疊到彼此附近,它們更傾向位於細胞核的哪些區域;以及當細胞狀態變化時——比如在DNA複製的不同階段——這些基因之間的相互作用關係又會如何隨之重排。
4D是如何構建的
研究人員們選擇了兩種具有代表性的細胞種類,是人體中細胞發育的起點和終點:未經分化的人胚胎幹細胞H1-hESC,和到達發育終點的永生化成纖維細胞HFFc6。
識別染色體的技術在20多年前,由項目中的一組研究人員發明出來,這種技術的目的是測量酵母中少量DNA序列之間的接觸頻率。爲了能用在染色體4D圖譜計劃中,研究人員對這種方法進行了大量的優化,使其可以測量更大的規模和更高的分辨率。
研究人員用四步抓到染色體環。
第一步:固定
研究人員先用化學試劑處理細胞,像是處理新鮮動物標本時的防腐步驟一樣,把當時染色質的空間狀態固定下來,避免在後續操作中散開。
第二步:剪碎
接着用“剪刀”把染色體剪成許多小片段。一般來說,剪得越細,後面定位越精確。
第三步:就地拼接
在DNA被剪開的狀態下,加入連接酶讓這些片段進行就地拼接。當時在細胞核裏離得越近的片段,越容易被粘到一起。
第四步:測序讀取
最後對這些拼接產物進行測序。研究人員會讀到一種關鍵證據:一條序列的前半段來自基因組的A位置,後半段卻來自很遠的B位置——這說明A和B在細胞核裏曾經非常接近。在大量這樣的證據彙總後,我們所說的染色質環就被抓住了。
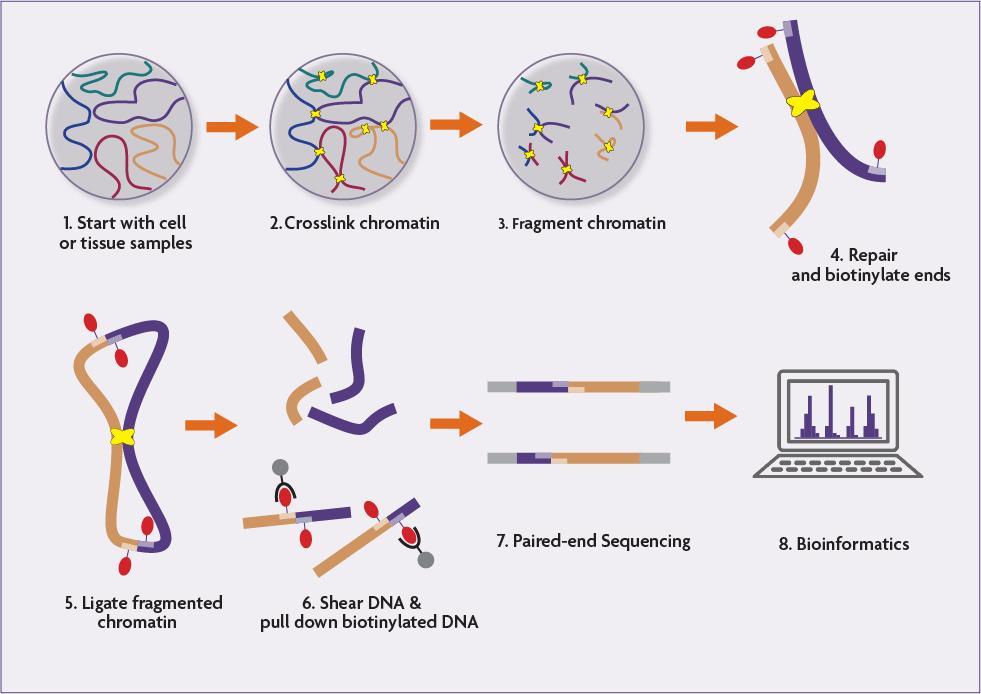
染色質環測序核心流程——固定、剪切、連接、測序讀取,本圖展示了Hi-C測序法。但無論什麼方法,核心流程是不變的|論文
3D基因組的測序方法不止一種,而每種方法擅長捕捉的環類型是不一樣的,論文中,研究人員使用了7種測序方法,儘可能地全面捕捉染色質環。
在獲得原始數據後,研究人員還要像後廚洗菜一樣,對數據進行進一步的清洗、定位。篩掉重複和低質量片段,以及把得到的片段定位回基因組。
接下來纔是最關鍵的一步:從海量的配對信息裏找出真正可靠的信息。研究人員會進一步篩選那些在統計上足夠突出、在不同重複中也足夠穩定的基因位置,只有滿足這些條件的,纔會被認定爲可靠的染色質環。
經過這樣一輪輪大浪淘沙、精益求精的篩選,研究團隊最終在兩種細胞中分別構建出規模驚人的環目錄:每種細胞各自識別並編目了約14萬條染色質環數據。
藉助這些龐大又精確的數據,研究人員不僅能更完整地描繪某個基因所處的染色體環境,還可以推斷它可能與哪些遠端調控元件或其他基因發生互相作用,並進一步把關鍵遺傳過程放回到染色質的立體結構中進行定位與理解。
4DN模型可以直接幫助我們將細胞的結構對應到相應的功能|論文
之後,就該搭建第四維了。
這裏的時間,並不是我們直覺裏那種從生到死給細胞拍一段連續視頻,在這項工作中,4DN主要用兩種方式把時間引入圖譜。
第一種,是以細胞週期中的DNA複製作爲天然的時間軸——DNA 複製本身就有明確的先後順序。研究人員將三維結構特徵與複製時序數據逐一對應,觀察不同摺疊狀態與複製的進程,以及基因激活等過程之間的對應關係,從而把靜態的三維結構放回到細胞週期推進的時間框架中理解。
第二種,是從單細胞差異入手,把變化當作動態的一部分:通過比較不同細胞類型之間的差異,以及同一種細胞內部個體之間的波動範圍,研究人員得以判斷哪些結構特徵相對穩定、是一定會保留的看家基因;哪些更靈活,會隨着細胞功能狀態或週期階段的變化而變化。
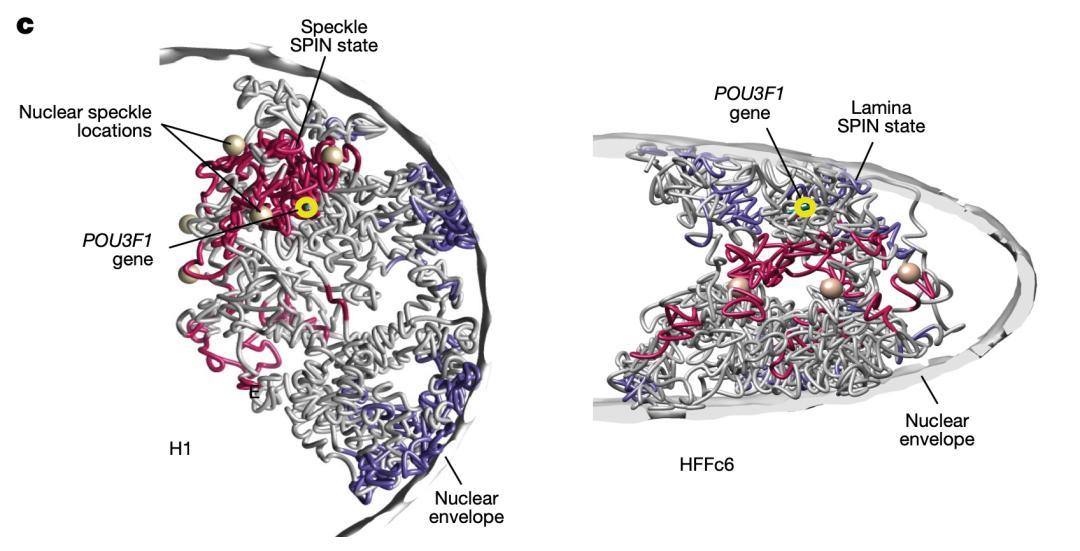
細胞間差異:POU3F1基因在胚胎幹細胞中被沉默(紅色),在成纖維細胞中激活(灰色)|論文
4DN圖譜有了,下一步呢?
染色質結構的異常與先天性出生缺陷和癌症發展密切相關。理解DNA的動態4D組織,將幫助研究人員弄清核內結構的變化會影響哪些基因被打開或關閉,以及異常的核組織如何打斷正常的發育與細胞功能,從而引發人類疾病。
例如在EPHA4基因附近,存在着特定的結構來限制增強子的作用範圍、精確調控基因表達。一旦某些結構變異或突變破壞了這套機制,原本只負責驅動EPHA4的增強子就可能串臺,誤觸發隔壁的發育基因,進而導致不同程度的肢體發育畸形,例如多指、並指等。
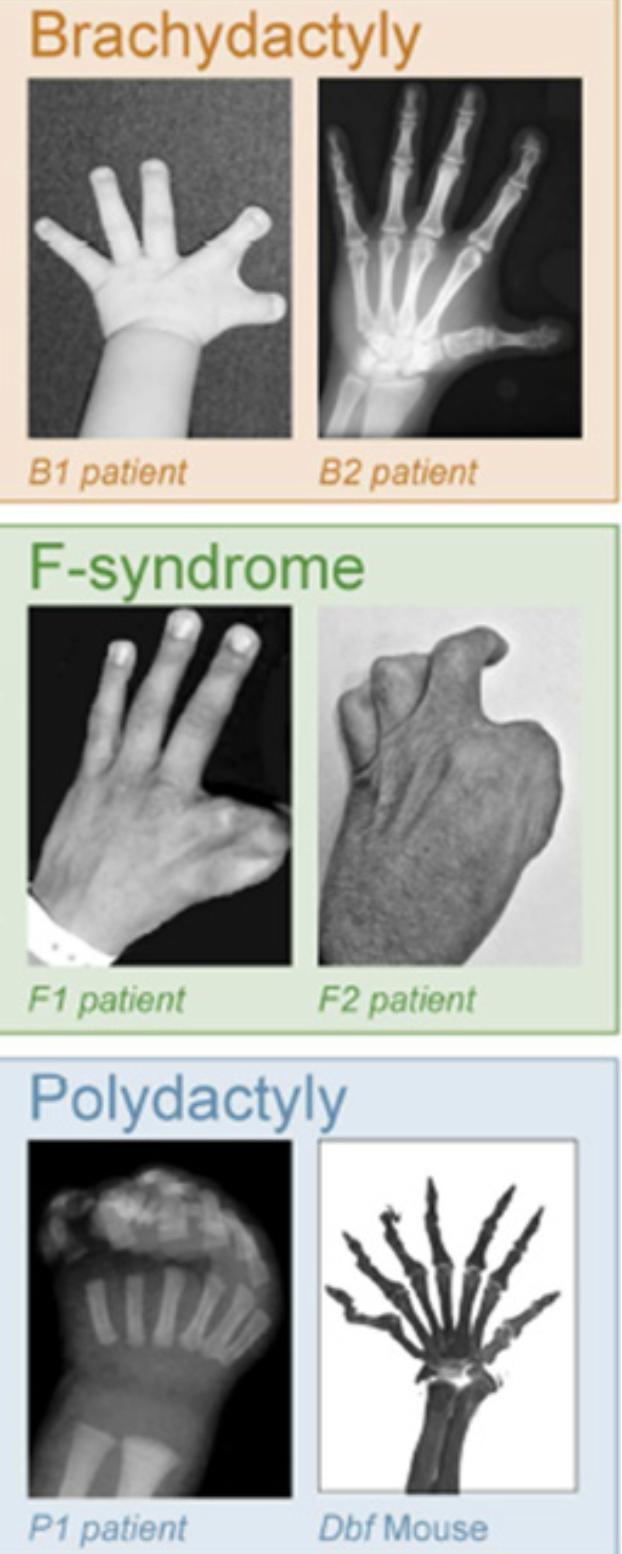
EPHA4結構異常導致的畸形 |文獻
4DN計劃正在努力使生物醫學研究界能夠確定治療由異常核組織引起的人類疾病的新目標。可能有更多原因未知的疾病,可以通過4DN來找到病因和治療手段。
因此,4DN計劃的目標不僅是建立圖譜,更可以讓生物醫學研究界能夠據此找到由異常核組織引起疾病的新靶點。未來,許多目前病因不明的疾病也許能借助4DN的框架被重新解釋——從結構層面找到真正的致病環節,併爲更精準的診斷與治療提供線索。
在繪製4DN圖譜的過程中,也誕生了不少非常實用的“副產品”。要把來自不同團隊、不同平臺的大量數據匯聚成一個可用的公共資源,標準化和流程化是不可少的——就像現代工業生產不可能再像小作坊一樣一件件手搓。
因此,在過去十年裏,4DN項目組逐步推動並完善了測序數據的統一標準與格式,使書同文,車同軌,確保每個實驗室產出的數據格式一致且通用。這樣一來,不同來源的數據能夠更順暢地拼接、對照,並最終匯入同一張圖譜。這種標準化不僅打破了實驗室間的數據壁壘,還促成並加速了不同地區之間的合作,是一項功在當代、利在千秋的舉措。
同時,4DN在持續更新和維護項目論文列表與產出集合,使方法學、數據資源、工具等都保持最新,讓全球的研究人員們都能以更高效的方式來完成研究。在逆全球化情緒抬頭的當下,4DN這種開放共享、沒有國界的協作方式,確實讓人精神一振。
光建成數據庫還不夠,研究人員更想把它用起來。下一階段,4DN的目標不再只是給出一張染色體地圖,而是要把這張圖譜落到每一個細胞上,讓我們能夠讀出細胞在某一時刻的實時生理活動。
爲此,4DN圖譜需要將兩類單細胞數據聯動:一類是高分辨率顯微鏡下的單細胞成像數據,直接看到染色體與核內結構在空間中的位置;另一類是單細胞的基因組學數據,讀出同一個細胞裏基因表達、複製等分子事件的狀態。把這兩類信息整合起來,研究人員就有機會從靜態走向動態,還原細胞當下的實時狀態。
這些努力最終要回答一個簡單,但是核心的生物學問題:每個細胞結構、功能的不同導致每個細胞核都不完全一樣,甚至差異很大,但生命體卻能穩定發育、穩定運行。那麼,細胞究竟是如何在這種千差萬別的背景下,仍然可靠地執行同一套基因表達程序,並把結果控制在可控、可預測的範圍內?
胎兒的發育,很神奇吧 |來源:GIPHY
如果真的能夠回答這個問題,那麼人類就將理解在人體這個如此混沌的系統中,不同的細胞是如何各顯神通、互相合作,保持整個系統的平衡。我們也能反過來利用這點,將因爲受傷、得病、衰老而失衡的人體重歸於平衡之中。
參考文獻
[1] Lupiáñez DG, Kraft K, Heinrich V, Krawitz P, Brancati F, Klopocki E, Horn D, Kayserili H, Opitz JM, Laxova R, Santos-Simarro F, Gilbert-Dussardier B, Wittler L, Borschiwer M, Haas SA, Osterwalder M, Franke M, Timmermann B, Hecht J, Spielmann M, Visel A, Mundlos S. Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions. Cell. 2015 May 21;161(5):1012-1025. doi: 10.1016/j.cell.2015.04.004. Epub 2015 May 7. PMID: 25959774; PMCID: PMC4791538.
[2] Dekker, J., Oksuz, B.A., Zhang, Y. et al. An integrated view of the structure and function of the human 4D nucleome. Nature (2025). https://doi.org/10.1038/s41586-025-09890-3
[3] https://www.activemotif.com/catalog/1317/hi-c-service
作者:李小雅
編輯:翻翻
封面圖來源:Nature
Guokr
24節氣中最後也是最冷的一個節氣——大寒要來了。主打科技保暖的素湃,放出一波冬季重磅福利!
明早7點,除了抗寒衣,保暖內衣、衛褲等等,全場59元起,給你從頭到腳的禦寒武裝。就趕快預約直播!
本文來自果殼,未經授權不得轉載.
如有需要請聯繫[email protected]
點個“小愛心”吧