長頸鹿有四種?如何從1到4,解開物種分化的謎題
長頸鹿可能是動物園中的明星物種了,每個參觀長頸鹿的人都會驚歎它們的長脖子。在我們普通人看來,所有的長頸鹿似乎都是一樣的——長脖子,花斑紋。
但最近世界自然保護聯盟(IUCN)發佈了一份里程碑式的評估報告,正式確認:長頸鹿不止一個物種而是四個物種,包括北方長頸鹿,南方長頸鹿,網紋長頸鹿和馬賽長頸鹿。其中一部分的區分依據就是它們身上的條紋顏色以及分佈區域。比如北方長頸鹿和網紋長頸鹿就分佈於非洲偏中部和東北部。

蘇州上方山動物園的網紋長頸鹿
(圖片來源:作者拍攝)

南方長頸鹿,如果仔細對比,我們會發現它們二者的花紋是有區別的
(圖片來源:作者拍攝於天津動物園)
當然,要IUCN承認長頸鹿分爲四個物種,並不只是依靠長頸鹿身上的斑紋不一樣這一個證據這麼簡單。事實上,現代科學中要區分物種還挺複雜的——雖然我們從小在生物學中就瞭解了生殖隔離,但生殖隔離並不是區分物種的金標準。
一、爲什麼不能僅根據生殖隔離劃分物種?
在最初的分類實踐中,生物學家普遍把“能不能生出可育後代”當作物種分類的標準,這就是課本里常見的“生物學物種概念”(BSC,Biological Species Concept)。按照這個思路,兩個羣體如果交配後代大多不育,就說明它們之間存在生殖隔離(不同羣體難以穩定產生可育後代)。
比如我們熟知的和驢子生下來的馬騾和驢騾(前者是父親爲驢,後者是父親爲馬,最常說的騾子是馬騾,),就是幾乎沒有生殖能力的,即使偶有生殖,但從羣體來看也是並不穩定的。或者是雜交物種的某一個性別不具備生殖能力,比如雄獅和雌虎生下來的獅虎獸則是雄性能力不會生,但是雌性則有重新和雄獅、雄虎生娃的案例。還有黃牛和家犛牛生下來的犏牛,也是雄性不能生,雌性可育繼續生育。這些經典例子讓BSC顯得既直觀又好用。

獅獅虎獸(雄獅和雌獅虎獸所生)
(圖片來源:Wikipedia)
但是問題在於,大自然並沒有這麼簡單。科學家就發現了許多不同物種生下來的後代依舊能繁殖的例子。比如在北極圈附近,就有棕熊和北極熊雜交然後生下來的“棕白熊”,這些熊依舊有生殖能力。在海豚科下面,一些不同物種的海豚之間甚至可以形成穩定的雜交羣體。此外,北美洲的郊狼和灰狼也時常誕下具有生殖能力的後代。因此,如果我們教條地按照能不能“生出可育後代”這個標準劃分物種的話,我們就會把許多原本各自獨立演化的譜系合併到一起了。

棕熊和北極熊混血個體
(圖片來源: TreeHugger)
因此,現代分類學對物種的分類採取了非常謹慎的態度。不僅會考慮到是否存在生殖隔離,還會考慮形態特徵、地理與生態分佈等多種因素,只有當多個因素指向同一個結論時,我們纔會明確物種的劃分。
二、物種到底要如何劃分?
在多種劃分物種的依據中,形態學無疑是最直觀的證據。科學家首先會看骨骼、牙齒、體色與花紋等是否在不同羣體中穩定可辨,這要求在同一地區、不同個體之間都能反覆識別,儘量排除環境造成的可塑性差異。比如印度犀與爪哇犀,就在頭骨與門齒形態上有系統性區別。我們在前文提到的北極熊和棕熊雖然僅僅分化才幾十萬年,但它們卻形成了不同的頭骨形態、臼齒、毛色和耳朵大小等,這也是將它們分爲兩個物種的依據。
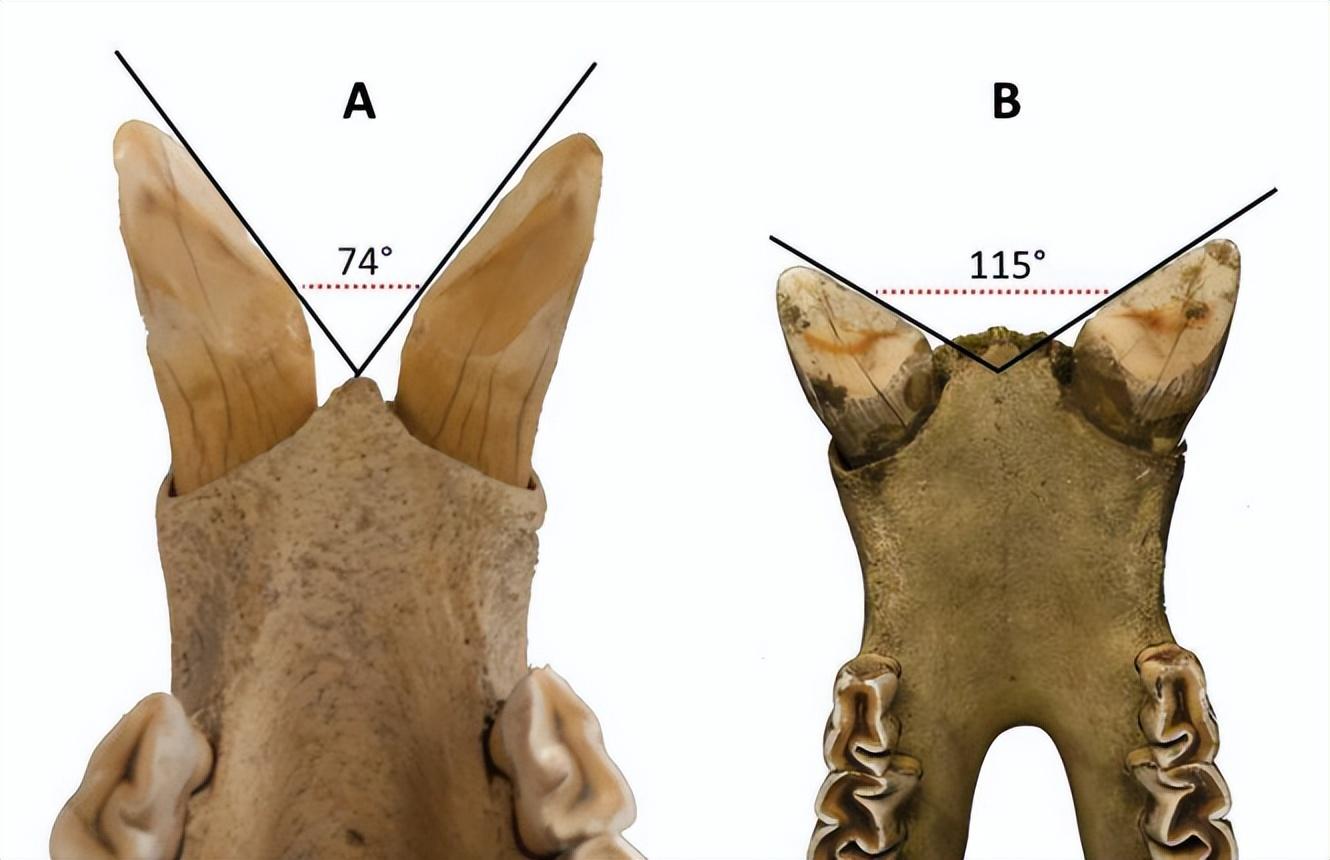
左側爲爪哇犀的門齒,右側爲印度犀
(圖片來源:參考文獻4)
遺傳學就是深層證據之一。研究者會比較不同羣體的DNA,看它們是否已經出現了長期的遺傳分化並考察羣體間的基因交換是否持久偏低。這裏常用到核基因組和線粒體基因組的搭配:前者來自父母雙方,能反映總體譜系關係;後者只隨母系傳遞,能幫助識別歷史上的雌系軌跡。以非洲大象爲例,就被分爲草原象與森林象,前者具有較大的耳朵和體重,不一樣的頭骨以及門齒形態,數量也比起後者多很多,分佈區域也存在一定差別,但它們在覈基因組上分化也非常顯著,線粒體譜系也長期分開,說明兩者不只是外形不同,而是在演化史上各走各路。遺傳證據的意義在於,它能跨過短期的環境影響,給出時間尺度更長的分家賬。

非洲草原象,拍攝於上海自然博物館
(圖片來源:作者)
當然,科學家也會考慮生殖層面的證據。但是現代研究更關心的是羣體是否能長期維持獨立,而不是單個雜交事件是否存在。雖然在前文提到的棕熊和北極熊、海豚、郊狼和灰狼等之間確實存在可育雜交的記錄,但這些現象多出現在接壤狹窄地帶或特定生態窗口,比例低,很難在大範圍內把兩個物種的基因庫融合到一起。這些生殖隔離很模糊的不同物種,則會被統稱爲“物種複合體”,也稱之爲分類複合種(species complex)。它們形態學上有不少類似,親緣上有些錯綜複雜,以至於難以確認種間界限。
當然,有時候兩個物種之間的雜交種在長期演化之後,也會成功產生新的物種。比如俗稱爲狗熊的亞洲黑熊就是由熊科的兩個不同支系混血雜交之後形成的新物種。還有黔金絲猴也是川金絲猴和其他金絲猴屬支系混血而成,毛色繼承了川金絲猴支系的黃色毛髮也有其他金絲猴屬的黑色毛。還有北美洲由郊狼和灰狼混血產生的赤狼(Canis rufus)這個物種(只分布在美國南部及其附近,極危物種)。

黔金絲猴,拍攝於北京動物園
(圖片來源:作者)
最後還要看地理與生態的版圖。不同羣體如果長期分佈在不同區域,被山脈、沙漠、洋流或氣候帶分隔,或者佔據了不同的生態位,它們在行爲、食性與微環境選擇上就會逐漸拉開差距。這樣的差距會反過來進一步降低基因交流,最後形成不同物種。在長頸鹿身上,我們看到不同譜系分佈在非洲的不同地帶,棲息偏好與活動範圍並不相同,這些地理與生態差異與前述的形態和遺傳證據一起,構成了同一個故事的多個側面。
三、物種亞種的分化
但有時候,即使使用了這些綜合證據,但不同證據之間還會存在不一致的情況,這是因爲分化時間短或者生殖隔離不完全之類的原因,此時研究者就會使用“亞種”來記錄分化。它與物種的區別在於:基因交流並未完全中斷,遺傳分化的深度較淺,外形差異雖然穩定,但不足以支撐“長期各自演化”的結論。多數亞種呈現地理分佈上的分割,彼此之間常有過渡帶,也就是在鄰接區域能看到漸變或雜交個體)這說明它們仍屬於同一物種的不同地域型。
例如我們熟悉的金錢豹就在IUCN上被列爲8個亞種,包括非洲亞種(指名亞種),中國東北和華北,以及川陝甘等地的遠東亞種,印度次大陸和斯里蘭卡的亞種等。老虎也屬於同一物種的多亞種格局:東北虎。華南虎,孟加拉虎等被認爲起源於約7-11萬年前的共同祖先,隨後在氣候變化與棲息地分化的背景下逐漸形成了各自穩定的頭骨與被毛差異,這些差異在地域上能反覆識別,但並未完全達到不同物種的門檻。

華南虎,毛色深一些且較短,拍攝於上海動物園
(圖片來源:作者)

毛色淺一些且較爲蓬鬆的東北虎,拍攝於長春動植物公園
(圖片來源:作者)
但也有物種在討論“要不要劃亞種”的同時,發現內部已形成長期獨立的譜系,但形態差異並不顯著,因而尚未正式命名亞種。海洋中的大白鯊就是一個例子:基因組研究顯示,其在十幾萬年前已分化爲北太平洋、北大西洋和印度洋—太平洋三大譜系,這些譜系幾乎互不通婚,但目前仍作爲同一物種下的不同遺傳支系來討論,等待更多證據明確是否需要在命名層級上作進一步處理。
四、爲什麼要定好物種和亞種?
分類並不是書齋裏的命名遊戲,因爲無論是研究還是保護,其資源都是有限的。把物種的邊界釐清之後,哪一個物種更稀少,哪一個物種是關注重點,哪一些個體可以配對等問題就能更清晰了。更簡單地來說,先認清楚不同物種的譜系,再避免外來血緣對本地物種的基因污染,這樣保護和繁育才不會南轅北轍。
由物種不清導致的保護問題中的一個例子是全球只剩兩隻的一級保護動物斑鱉,這兩隻斑鱉均確認爲雄性。斑鱉如此稀少,瀕臨滅絕,與它們在過去經常因爲名字和分類地位,被和國家二級保護動物黿搞混有關係。

蘇州上方山動物園的斑鱉蘇蘇,全球僅剩下確認的斑鱉之一
(圖片來源:作者)
回到文章開頭的長頸鹿。這四種長頸鹿在分佈區域、骨骼形態、核基因組和線粒體基因組等都存在較多的差別。且不同種類的長頸鹿的個體數量也不相同,其中分佈在中非共和國,南蘇丹等國家地區的北方長頸鹿數目只剩下7000多頭,南方長頸鹿數目則超過6萬頭,而網紋長頸鹿和馬賽長頸鹿在各自分佈區也面對不同的生境破碎化與人爲壓力。現在,新的物種分類在更新後,配額、走廊建設、種羣間調運與配對計劃都會按物種分別制定,而不再用同樣的標準來保護所有長頸鹿。這就能避免把資源過度分散導致的保護失調的問題。
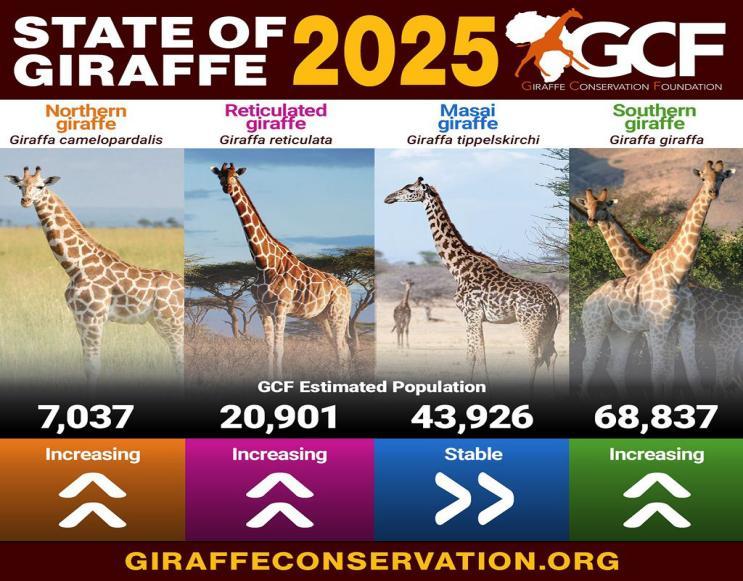
四種長頸鹿目前的數目,從左到右依次是北方長頸鹿,網紋長頸鹿,馬賽長頸鹿和南方長頸鹿。其中馬賽長頸鹿數目相對穩定,其他三種在增長
(圖片來源:giraffeconservation)
參考文獻:
- Julian Fennessy et al. Multi-locus Analyses Reveal Four Giraffe Species Instead of One. Current Biology.2016; 26(18): P2543-2549.
- Li HM, Liu BY, Shi MH, et al. Haplotype-resolved and chromosome-scale genomes provide insights into co-adaptation between the Amur tiger and Amur leopard. Zool Res. 2024;45(3):464-467.
- Wang MS, Murray GGR, Mann D, et al. A polar bear paleogenome reveals extensive ancient gene flow from polar bears into brown bears. Nat Ecol Evol. 2022;6(7):936-944.
- Nardelli F, Heißig K. A taxonomic review of the genus Rhinoceros with emphasis on the distinction of Eurhinoceros (Perissodactyla, Rhinocerotidae). Zookeys. 2025;1230:303-333.
- Broggini C, Cavallini M, Vanetti I, Abell J, Binelli G, Lombardo G. From Caves to the Savannah, the Mitogenome History of Modern Lions (Panthera leo) and Their Ancestors. Int J Mol Sci. 2024;25(10):5193.
- Amat F, Escoriza D. Biogeographic Inferences on the Evolutionary History of the King Cobra (Ophiophagus hannah, Cantor 1836) Species Complex. Zool Stud. 2022;60:e28
- Wagner I, Smolina I, Koop MEL, et al. Genome analysis reveals three distinct lineages of the cosmopolitan white shark. Curr Biol. 2024;34(15):3582-3590.e4.
- Das, Indraneil ; Gowri Shankar, P. ; Swamy, Priyanka et al. / Taxonomic revision of the king cobra Ophiophagus hannah (Cantor, 1836) species complex (Reptilia: Serpentes: Elapidae), with the description of two new species. In: European Journal of Taxonomy. 2024;961:pp.1.
- Wu H, Wang Z, Zhang Y, et al. Hybrid origin of a primate, the gray snub-nosed monkey. Science. 2023;380(6648):eabl4997.
- Zou T, Kuang W, Yin T, et al. Uncovering the enigmatic evolution of bears in greater depth: The hybrid origin of the Asiatic black bear. Proc Natl Acad Sci U S A. 2022;119(31):e2120307119.
- Wang MS, Wang S, Li Y, et al. Ancient Hybridization with an Unknown Population Facilitated High-Altitude Adaptation of Canids. Mol Biol Evol. 2020;37(9):2616-2629.
- Low WY, Tearle R, Liu R, et al. Haplotype-resolved genomes provide insights into structural variation and gene content in Angus and Brahman cattle. Nat Commun. 2020;11(1):2071.
- Wilkins JS. What is a species? Essences and generation. Theory Biosci. 2010;129(2-3):141-148.
- Curry CJ, Davis BW, Bertola LD, White PA, Murphy WJ, Derr JN. Spatiotemporal Genetic Diversity of Lions Reveals the Influence of Habitat Fragmentation across Africa. Mol Biol Evol. 2021;38(1):48-57.
- Bertola LD, Jongbloed H, van der Gaag KJ, et al. Phylogeographic Patterns in Africa and High Resolution Delineation of Genetic Clades in the Lion (Panthera leo). Sci Rep. 2016;6:30807.
- Rong RH, Cai HD, Yang XQ, Wei J. Fertile mule in China and her unusual foal. J R Soc Med. 1985;78(10):821-825.
- Gaughran SJ, Gray R, Ochoa A, et al. Whole-genome sequencing confirms multiple species of Galapagos giant tortoises. Evolution. 2025;79(2):296-308.
- Alcaide M, Scordato E S,Price T D, et al. Genomic divergence in a ring species complex. Nature,2014, 511(7507):83-85.
- Mondol S, Moltke I, Hart J, et al. New evidence for hybrid zones of forest and savanna elephants in Central and West Africa. Molecular ecology, 2015, 24(24): 6134-6147.
- Du H, Yu J, Li Q, Zhang M. New Evidence of Tiger Subspecies Differentiation and Environmental Adaptation: Comparison of the Whole Genomes of the Amur Tiger and the South China Tiger. Animals (Basel). 2022;12(14):1817.
- Liu YC, Sun X, Driscoll C, et al. Genome-Wide Evolutionary Analysis of Natural History and Adaptation in the World''s Tigers. Curr Biol. 2018;28(23):3840-3849.e6.
- Pilot, M. Disentangling the admixed trails of grey wolf evolution. Molecular Ecology.2021;30, 6509–6512.
- Hai-Meng Li, Bo-Yang Liu, Min-Hui Shi, Le Zhang, Shang-Chen Yang, Sunil Kumar Sahu, Liang-Yu Cui, Shan-Lin Liu, Nicolas Dussex, Yue Ma, Dan Liu, Wei-Yao Kong, Hao-Rong Lu, Yue Zhao, Love Dalén, Huan Liu, Tian-Ming Lan, Guang-Shun Jiang, Yan-Chun Xu. Haplotype-resolved and chromosome-scale genomes provide insights into co-adaptation between the Amur tiger and Amur leopard. Zoological Research.2024;45(3): 464-467.
- Chai J, Lu CQ, Yi MR, et al. Discovery of a wild, genetically pure Chinese giant salamander creates new conservation opportunities. Zool Res. 2022;43(3):469-480.
- Yan F, Lü J, Zhang B, et al. The Chinese giant salamander exemplifies the hidden extinction of cryptic species. Curr Biol. 2018;28(10):R590-R592.
- Nie WH, Wang JH, Su WT, et al. Species identification of crested gibbons (Nomascus) in captivity in China using karyotyping- and PCR-based approaches. Zool Res. 2018;39(5):356-363.
- Roca AL. African elephant genetics: enigmas and anomalies. J Genet. 2019;98:83.
- Roca AL, Ishida Y, Brandt AL, Benjamin NR, Zhao K, Georgiadis NJ. Elephant natural history: a genomic perspective. Annu Rev Anim Biosci. 2015;3:139-167.
出品:科普中國
作者:呂澤龍(中國科學院動物研究所)
監製:中國科普博覽