破解百年難題:AI設計出抗蛇毒蛋白
傳統抗蛇毒血清需要數月時間、依賴動物免疫,而如今,諾獎團隊開發的AI“蛋白質設計師”只需幾秒鐘的時間去定製設計一款蛋白。這項突破不僅讓抗蛇毒血清的耐熱性突破78℃、成本直降90%,更預示着人類正式進入“AI造藥”新時代。
蛋白質有多少種?比宇宙原子還多!
蛋白質是生命活動的“全能選手”,從細胞結構到免疫防禦,全靠它撐場子。雖然由20種氨基酸通過肽鍵連接形成,但排列組合方式多到爆炸。如果考慮所有可能的氨基酸排列方式,理論上,蛋白質的種類幾乎是無限的。僅100個氨基酸的鏈條,就能拼出20^100種可能,比宇宙原子總數還多!
蛋白質的種類幾乎是無窮的,但它們都由20種基本氨基酸按照不同順序和組合方式構成。生物體內的蛋白質種類受基因編碼的限制,例如人類基因組大約編碼2萬種蛋白質,而通過剪切、翻譯後修飾等機制,最終可能形成幾十萬種功能各異的蛋白質。不同物種、不同細胞類型甚至相同細胞在不同狀態下,都可能表達出不同的蛋白質組合。
蛋白質設計就是利用科學方法讓氨基酸以不同的排列方式“定製”蛋白質,使其具備特定的結構和功能。就像用不同的積木搭建出獨特的模型,科學家根據需求排列組合氨基酸,從而創造新的蛋白質。藉助計算機模擬和人工智能,我們可以更精準地預測和優化蛋白質結構,讓它們更穩定、更高效地完成目標任務。
最近科學家通過AI設計抗蛇毒蛋白,僅用幾秒鐘就可以完成人類百年抗蛇毒的問題。
傳統抗蛇毒血清:慢、貴、難!
據WHO統計,全世界每年有540萬人被蛇咬傷,約13萬人死於蛇咬傷,因蛇咬傷截肢等造成殘疾的人數大約是39萬人。蛇毒中的毒性成分進入人體後,會迅速對人體的神經系統、心血管系統、凝血系統以及各類細胞等造成損傷。

全世界每年約有540萬人被蛇咬傷
(圖片來源:pixabay)
正常情況下,免疫系統對於曾經接觸過的病原體能快速做出反應,但絕大多數人在被蛇咬之前並沒有接觸過蛇毒,所以,被蛇咬後人體自身的免疫防禦很難馬上起效,往往需要藉助外部的抗蛇毒血清等治療手段來儘快中和蛇毒,避免蛇毒對身體造成更嚴重的傷害。然而,世界上許多地區都存在嚴重的抗蛇毒血清不足。

收集蛇毒
(圖片來源:pixabay)
傳統血清怎麼來?首先,需要從毒蛇體內提取出毒液,再將少量毒液注射到馬或羊等動物的體內,誘導免疫反應的產生。經過多次免疫後,再從動物體內採集血液,分離出血清,即可獲得有效的抗蛇毒血清。一套流程下來,耗時數月,成本高昂,同時不同地區的蛇種不同,毒液成分差異大,傳統血清難以覆蓋所有蛇毒。
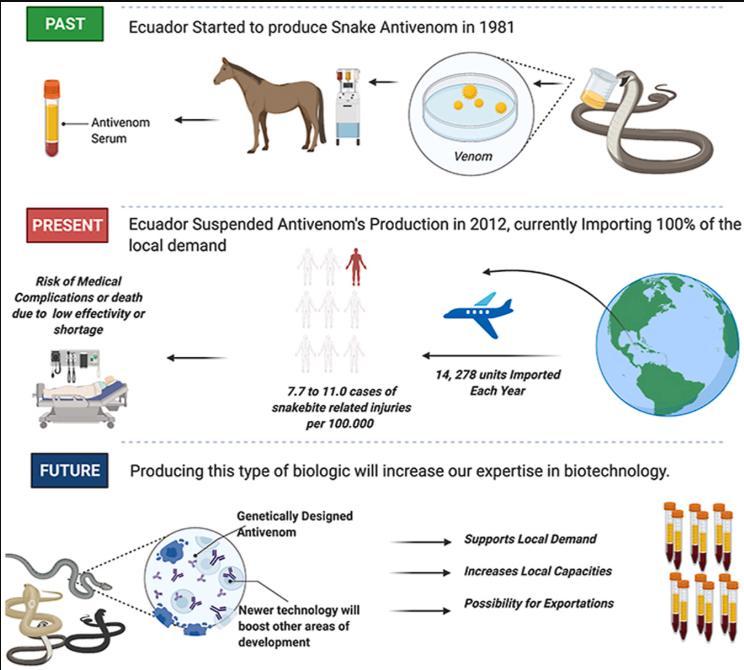
傳統的抗蛇毒血清製備過程
(圖片來源:參考文獻1)
AI出手,抗蛇毒血清更加有效
近年來,針對傳統抗蛇毒血清製備方法的改進研究取得了顯著進展,尤其是人工智能(AI)與計算機輔助設計技術的融合爲抗蛇毒血清研發帶來了新的突破。
2025年,美國華盛頓大學蛋白質設計研究所的David Baker 團隊在《自然》(Nature)雜誌發表重磅研究。他們開發的RFdiffusion深度學習方法,可以模擬出對一種廣泛存在於蛇毒中的小型蛋白毒素——三指毒素,具有高親和力和高特異性的結合蛋白。另外,通過計算設計出的抗蛇毒蛋白具有高熱穩定性,可通過微生物發酵策略大規模生產,和傳統血清製備技術比起來,極大地降低了生成成本。

發表在《自然》雜誌上的研究
(圖片來源:文獻3)
RFdiffusion可以生成具有特定功能的蛋白質結構,其工作原理可以分爲幾個部分:
1.數據訓練:通過分析大量已知蛋白質結構,模型掌握了蛋白質形成的規律。
2.結構生成:從隨機初始化的蛋白質狀態出發,逐步調整氨基酸殘基的位置,最終生成穩定的三維結構。
3.功能優化:通過多輪生成與優化,模型逐步調整蛋白質原子的位置,使其從最初的無序狀態演化爲符合生物學規律的穩定構象。

Diffusion模型的蛋白設計過程
(圖片來源:參考文獻2)
研究以α-眼鏡蛇毒素和IA型細胞毒素爲例,這兩種毒素均屬於三指毒素,其分子結構就像三支手指一樣從中央的核心區域伸展出來,但它們的長度、氨基酸構成和空間構象都不同,導致毒性機制和引發症狀各異。
α-眼鏡蛇毒素:主要作用於神經系統,阻斷神經信號傳遞,導致肌肉麻痹。
IA型細胞毒素:通過與細胞膜的脂質雙層相互作用,破壞細胞膜穩定性,引發組織壞死和炎症反應。
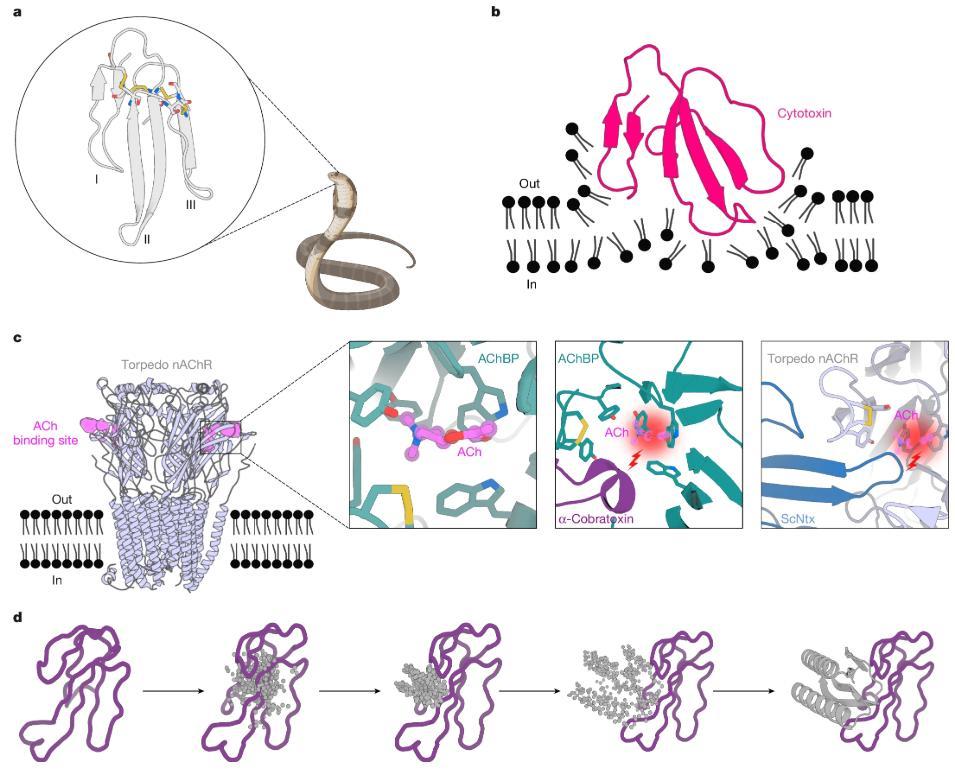
基於人工智能的抗血清蛋白設計思路
(圖片來源:參考文獻3)
那AI是如何精準設計抗毒蛋白呢?
在研究中,三指毒素的邊緣β-鏈是其與受體或抗體相互作用的關鍵部位,其表面氨基酸決定了毒素的特異性和親和力。比如,α-眼鏡蛇毒素的邊緣β-鏈結構,能精準結合人體菸鹼型乙酰膽鹼受體,阻斷神經信號傳遞。
研究團隊採用“分子互補”策略,以α-神經毒素和IA型細胞毒素爲目標毒素,使用RFdiffusion模型爲目標毒素的邊緣β-鏈設計出幾何互補的β-鏈蛋白,通過空間位阻阻止毒素與受體結合,這就像給一半殘缺的鏡子,通過科學手段做成互補的另一半。
具體來說,研究團隊將目標毒素的晶體結構輸入 RFdiffusion模型,模型依據輸入信息和學習到的蛋白質結構規律,調整氨基酸殘基的排列,促使生成的結構朝着與目標毒素β-鏈互補匹配,並形成穩定的β-摺疊的方向發展,最終構建出與目標毒素互補結合的蛋白質骨幹結構。這種設計使抗毒蛋白能緊密結合毒素,阻斷其與生理受體的相互作用,從而中和毒性。
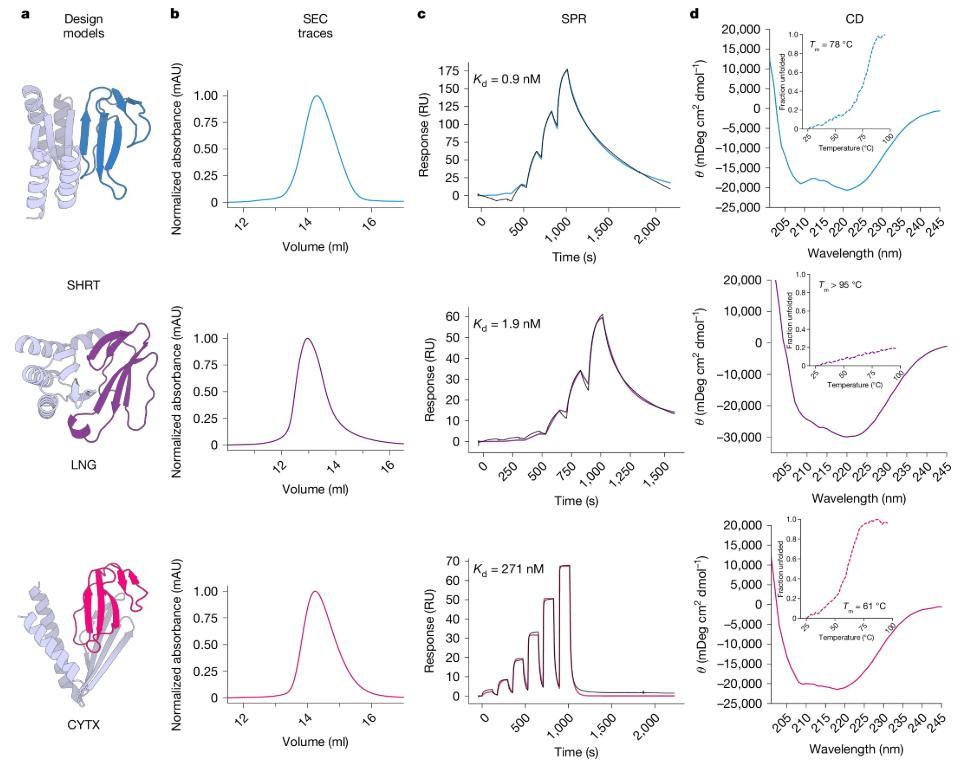
人工智能設計的抗血清蛋白的實驗性能表徵
(圖片來源:參考文獻3)
我們上面提到過,蛋白質的功能依賴於其特定的三維結構,而高溫會破壞其內部的氫鍵、範德華力等分子間作用力,導致蛋白質變性(如雞蛋加熱後凝固)。與傳統抗體相比,AI設計的抗毒蛋白展現出卓越的熱穩定性:耐受78°C以上高溫,遠超傳統抗體的耐熱極限。常溫下長期穩定,減少對冷鏈運輸和儲存的依賴,尤其適合熱帶地區使用。
蛋白質設計的演進:從理論奠基到AI革命
蛋白質設計領域經歷了從理論萌芽到人工智能驅動的跨越式發展,這一歷程堪稱現代生物技術的典範。
20世紀中期,Christian Anfinsen提出的"氨基酸序列決定蛋白質三維結構"假說,爲計算蛋白質設計奠定了理論基礎。隨着計算方法的進步,Rosetta軟件的問世標誌着蛋白質工程進入新紀元,該工具使科學家能夠在計算機上預測並設計穩定的蛋白質結構。2003 年,David Baker 團隊在《科學》(Science)雜誌發表的Top7蛋白研究具有里程碑意義,完全人工設計的蛋白質(98個氨基酸),並且在自然界中沒有同源蛋白。這項突破首次證明計算方法可以設計具有穩定摺疊結構的蛋白質,爲從頭重新蛋白質設計奠定了基礎。
人工智能的引入徹底改變了蛋白質設計領域。2020 年,DeepMind公司的 AlphaFold2在蛋白質結構預測領域取得歷史性突破,通過深度學習精確預測蛋白質結構,在 CASP14 競賽中達到了接近實驗的精度。隨後,Meta AI 的 ESMFold 進一步加速了蛋白質結構預測,實現未知蛋白質的大規模解析,爲蛋白質設計提供更豐富的數據基礎。同時,生成式 AI 的引入帶來質的飛躍,RFdiffusion 實現從頭設計功能性蛋白質,大幅提高了蛋白質工程的可塑性和效率。
2023 年,David Baker 團隊再創佳績,進一步推進人工智能在蛋白質設計領域的應用,開發了一種基於深度學習的 AI 算法,成功從頭設計出一種具有高催化活性和高底物特異性的人造熒光素酶。這是首次完全依賴 AI 生成全新酶類蛋白,標誌着人工智能驅動的蛋白質設計從結構預測邁向功能設計的關鍵轉折點。2024年,瑞典皇家科學院將諾貝爾化學獎授予David Baker,表彰其在計算蛋白質設計方面的貢獻,並將另一半授予Demis Hassabis和John M. Jumper,以表彰他們在蛋白質結構預測方面的突破。這一殊榮不僅是對個人成就的肯定,更是對整個蛋白質設計領域的認可,彰顯了計算生物學在現代科學中的核心地位。
AI 蛋白質設計正在突破傳統方法的侷限,大幅拓展蛋白質發現的邊界。與依賴天然模板或隨機突變的傳統方法相比,AI技術能夠突破進化限制,快速生成具有全新功能的蛋白質,如人工酶、智能生物材料等。通過深度學習和生成模型的結合,AI將蛋白質篩選與優化流程從傳統方法所需的數月時間,大幅縮短至數小時或數週。
在醫療應用方面,AI蛋白質設計展現出廣闊前景。除了前文提到的抗蛇毒血清外,這項技術還可開發抗菌、抗病毒蛋白及靶向降解工具,助力耐藥性疾病和神經退行性疾病治療。未來,AI蛋白質設計與自動化實驗技術的結合將帶來更多突破。這種融合將加速蛋白質功能優化過程,推動合成生命系統的構建,爲精準醫療、綠色能源和合成生物學等領域帶來革命性進展。
參考文獻:
- Ortiz-Prado, E., Yeager, J., Andrade, F., Schiavi-Guzman, C., Abedrabbo-Figueroa, P., Terán, E., Gómez-Barreno, L., Simbaña-Rivera, K., & Izquierdo-Condoy, J. S. (2021). Snake antivenom production in Ecuador: Poor implementation, and an unplanned cessation leads to a call for a renaissance. Toxicon : official journal of the International Society on Toxinology, 202, 90–97.
- Watson, J. L., Juergens, D., Bennett, N. R., Trippe, B. L., Yim, J., Eisenach, H. E., Ahern, W., Borst, A. J., Ragotte, R. J., Milles, L. F., Wicky, B. I. M., Hanikel, N., Pellock, S. J., Courbet, A., Sheffler, W., Wang, J., Venkatesh, P., Sappington, I., Torres, S. V., Lauko, A., … Baker, D. (2023). De novo design of protein structure and function with RFdiffusion. Nature, 620(7976), 1089–1100.
- Vázquez Torres, S., Benard Valle, M., Mackessy, S. P., Menzies, S. K., Casewell, N. R., Ahmadi, S., Burlet, N. J., Muratspahić, E., Sappington, I., Overath, M. D., Rivera-de-Torre, E., Ledergerber, J., Laustsen, A. H., Boddum, K., Bera, A. K., Kang, A., Brackenbrough, E., Cardoso, I. A., Crittenden, E. P., Edge, R. J., … Baker, D. (2025). De novo designed proteins neutralize lethal snake venom toxins. Nature, 10.1038/s41586-024-08393-x. Advance online publication.
出品:科普中國
作者:Denovo團隊
監製:中國科普博覽