這纔是 AI 近年來最有價值的成就,卻被很多人忽視
有一個英文單詞,它由 189,819 個字母組成。在常規語速下,要念完它需要花足足三個半小時——它是我們體內的肌聯蛋白(titin)的化學全名。
肌聯蛋白是人體內最大的蛋白質,由超過 3.4 萬個氨基酸組成。相比於只含幾百上千個氨基酸的常見蛋白質,肌聯蛋白實在是太大了。於是,科學家決定化用古希臘神話中巨人神族的名字“泰坦”(Titan),將它命名爲 titin。
然而,自科學家 1954 年發現肌聯蛋白已經過去 70 多年,我們依然不知道這位“巨人”的真實模樣。要用傳統的實驗方法解析出這個龐然大物的完整結構,幾乎是不可能的。

過去,想要知道某個蛋白質的全貌(三維結構),科學家只能依賴於核磁共振、X 射線晶體學或冷凍電鏡等手段。哪怕是一個只含幾百個氨基酸的蛋白質,科學家也得反覆表達純化樣本、測試條件,實驗室設備動輒上百萬美元,時間成本少則數月,多則幾年,就更別提肌聯蛋白這種擁有三百多個結構域的超級蛋白質了。如今,科學家才解析出肌聯蛋白極少部分的結構,距離拼出全貌還相當遙遠。
直到五年前,一款專門用於預測蛋白質結構的 AI 工具問世——在我們只是用 AI 洗稿、編論文、畫色圖之前。
太好了,是 AlphaFold
蛋白質結構幾乎決定了蛋白質的一切性質:蛋白質和誰互動,是否會引發疾病,以及哪裏可以作爲藥物作用的靶點。對科學家而言,蛋白質結構就像一張地圖,沒有這張地圖地科學家便只能摸黑走路,靠不斷嘗試來找到通往目的地的路徑。
可現實是,在浩瀚的蛋白質世界裏,人類真正通過實驗看清、畫出地圖的區域,只有極小的一部分。幾十年來,全世界科學家解析出的蛋白質結構還不到 20 萬個。而在所有人體蛋白質中,也只有約 35%的結構被實驗解析,且很多都殘缺不全。
蛋白質的結構由其氨基酸序列決定,我們能否直接通過氨基酸序列,預測出蛋白質的三維結構?這曾是生物學領域最艱難、最核心的挑戰之一。
但是,2020 年 11 月 30 日,AlphaFold2 出現了。
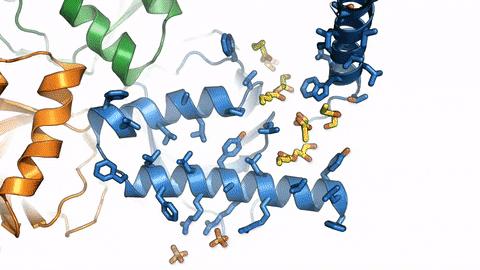
DeepMind 將蛋白質數據庫(PDB)中實驗得到的蛋白質結構“喂”給了神經網絡,讓 AI 學習序列與結構之間的關係。最終,他們做到了科學家過去無法想象的事:在那年的“全球蛋白質結構預測比賽”(CASP)中,AlphaFold2 憑藉氨基酸序列預測蛋白質結構的準確率超過了 90%,直逼實驗方法。
現在,AlphaFold 數據庫中的蛋白質預測結構已超過 2 億個,其中也包括人類蛋白質組 98.5%的結構。來自 190 多個國家的上百萬科研人員正利用這一開源“蛋白質 GPS”,在前所未有的高效下破解蛋白質本身的奧妙——隨之而來的真實改變,其實已經陸續在我們日常生活中發生。

關鍵時候它真救命啊
早在 AlphaFold2 正式發佈之前,它就在人類的危機關頭展示了威力。
2020 年 3 月,新冠疫情剛爆發不久,DeepMind 就利用仍在開發中的 AlphaFold2,預測出了新冠病毒部分蛋白質的結構。在那樣的關鍵時刻,等待數月甚至數年時間解析結構並不現實。儘管 AlphaFold2 給出的預測並非最終答案,但它讓研究者能迅速瞭解一種新型病毒的生理機制,甚至篩選出可能的治療藥物。

它還正在輔助科學家研發疫苗,阻止下一次疫情發生。例如,英國牛津大學的研究團隊用 AlphaFold2 找到了一種瘧原蟲蛋白質的關鍵結合位點。如果將這些位點製成疫苗,讓人體產生相應的抗體,那麼蚊子叮咬接種過疫苗的人後,這些抗體進入蚊子體內,就會阻止瘧原蟲在蚊子腸道內發育,從源頭切斷瘧疾傳播。
遺傳病能不能不遺傳?
正因爲蛋白質結構無比重要,因此當基因的錯義突變改變蛋白質氨基酸序列後,就可能影響蛋白質結構,進而可能引發疾病。如今,馬耳他大學的科學家正在使用 AlphaFold 來研究基因突變對骨質疏鬆的影響。他們會生成正常蛋白質和突變蛋白質的結構模型,對比觀察這些突變是否可能破壞蛋白質的功能。

然而,這只是滄海一粟。在人類基因組中,存在多達 7100 萬種可能的錯義突變,而其中 99.9%的變體都屬於意義不明,我們不知道它們是否會導致疾病。

於是 2023 年,DeepMind 又在 AlphaFold 的基礎上推出了新模型 AlphaMissense。它並不直接預測結構,而是利用 AlphaFold 對結構的“直覺”,來判斷某個錯義突變是否可能致病。通過這種方法,研究團隊成功歸類了人類所有錯義突變中的 89%,生成了一份“人類錯義突變目錄”。有了這份目錄,科學家就能更有針對性地鎖定罕見遺傳病甚至糖尿病等複雜疾病背後的遺傳因素。
塑料污染問題,也許還有救
AlphaFold2 的作用遠不止預測結構,它還能幫助科學家重新設計蛋白質,去完成過去很難做到的事,例如高效地降解一次性塑料。

在英國樸次茅斯大學,科學家正在從微生物中尋找具有降解潛力的酶。有了 AlphaFold,他們短短几天內就能得到上百種候選酶的三維結構。於是他們就能基於這些結構,設計反應更快、穩定性更高、成本更低的酶,從而更有效地分解塑料垃圾。
首款 AI 製藥
你在用 AI 畫色圖,而人家在用 AI 製藥。
例如,生物技術公司 Insilico Medicine 就將 AlphaFold2 整合進了他們的端到端 AI 藥物發現平臺 Pharma.AI。他們用生物計算引擎 PandaOmics 尋找疾病的關鍵蛋白質靶點,再用 AlphaFold 預測其結構,最後讓 AI 藥物設計引擎 Chemistry42 基於該結構來設計藥物。
使用這種 AI 全流程的方法,他們短短几年內就找到了一種有望治療特發性肺纖維化的候選藥物——Rentosertib。Rentosertib 不僅是全球首個完全由 AI 發現生物靶點並設計苗頭化合物的試驗藥物,而且目前已經進入 II 期臨牀試驗階段,真正走上了藥物開發的主舞臺。
AI 時代更真實的意義
回到這個故事的起點,肌聯蛋白。
儘管就連 AlphaFold2 也無法一次性給出肌聯蛋白的完整結構,但它將肌聯蛋白的序列切分成了 166 個“重疊羣”(contig),每個都長達上千個氨基酸,並分別給出了相應的結構預測。
如今,科學家已經將這些結構整合起來,得到了肌聯蛋白中所有相互串聯的雙結構域的三維藍圖。與此同時,他們還藉助 AlphaMissense 和其他工具,評估了這一人體最大蛋白質不同突變的潛在致病性,讓一個此前幾乎無法觸及的研究對象突然變得有跡可循。
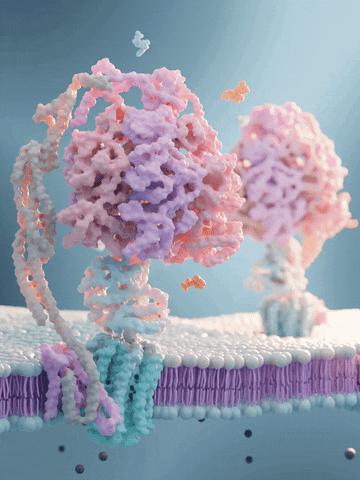
在 AlphaFold2 誕生後的這五年裏,生命科學本身並未停下腳步。2024 年,科學家又發現了一種比肌聯蛋白還要大 25% 的蛋白質——PKZILLA-1,刷新了自然界已知最大蛋白質的紀錄。
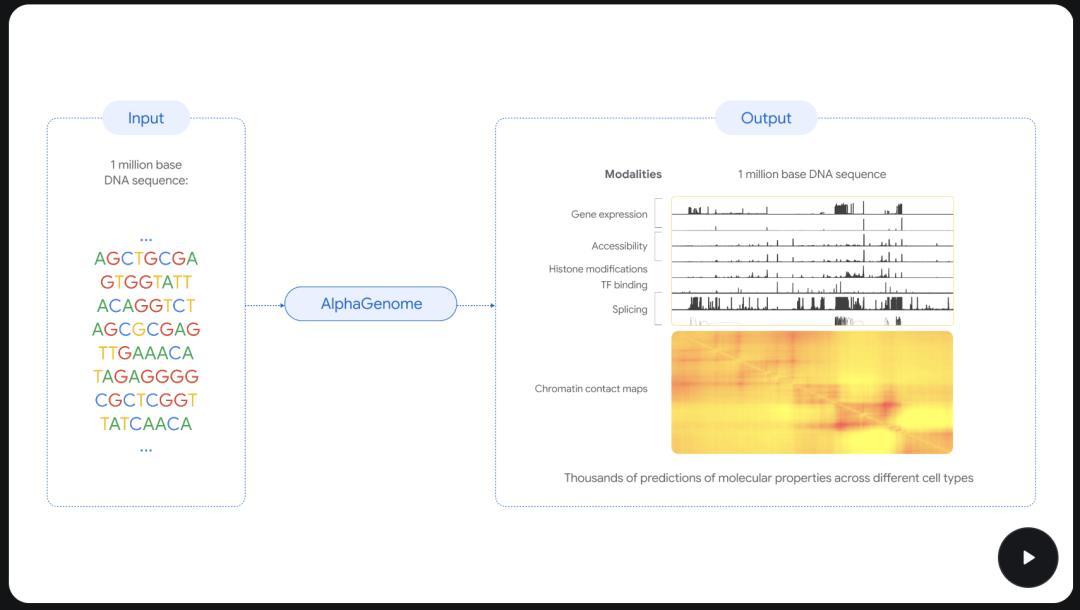
同年,谷歌也發佈了 AlphaFold3 和 AlphaProteo,前者能精準預測蛋白質與 DNA、RNA 等其他生命分子的相互作用,後者則能主動設計出能與疾病靶點結合的定製蛋白質。2025 年,谷歌又推出了 AlphaGenome,進一步把預測能力延伸到了 DNA 層面,從 DNA 序列預測基因突變對生物學過程的影響。
在蛋白質結構預測領域,AlphaFold 也並非獨行者。RoseTTAFold、ESMFold 和 I‑TASSER 等 AI 模型,也正在各自的道路上不斷突破,嘗試從不同角度解決困擾了生物學界幾十年的蛋白質摺疊問題。
在你偶爾會抱怨甚至厭煩滿眼 AI,甚至懷疑其泡沫屬性的時候,想想這些在最重要的科研領域真實發生的改變,就會更樂於迎接這個新時代的到來。
作者:黃雨佳
編輯:臥蟲
封面圖及插圖來源:Google、Giphy

Guokr
12月5日晚19點,果殼✖️六福珠寶黃金飾品福利專場來啦!
年底買金想撿漏一定要蹲住這場!給大家精選了40+款黃金飾品:項鍊、手鐲、手鍊、吊墜、戒指、串珠都有。全場專屬折扣價!下單還有超值滿贈、福袋。
大家一定要預約!!
本文來自果殼,未經授權不得轉載.
如有需要請聯繫[email protected]